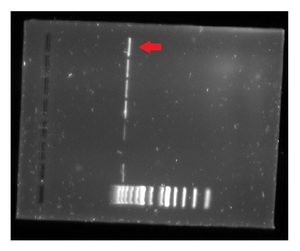
Das Gel
Das Gel (aus Agarose, daher Agarosegel) dient der Sichtbarmachung der DNA Fragmente. Die einzelnen Fragmente "laufen" durch das Anlegen von Strom durch ein Agarosegel und werden so nach Größe sortiert. Je größer ein Fragment ist, desto langsamer wandert es, da es sich langsamer durch die Strukturen des Gels bewegt als kleine Fragmente. Anschließend werden die DNA-Fragmente eingefärbt und unter UV-Licht sichtbar gemacht.
LAVES nutzt aktuelle Technik zur Insektenbestimmung
Verschiedene Arten werden mittels PCR, Sequenzierung und Restriktionsverdau ermittelt
Die Bestimmung von Insekten wird im LAVES traditionell mittels Mikroskop/Binokular vorgenommen. Allerdings können beschädigte Insekten und Insektenreste nur schwer bis gar nicht mikroskopisch bestimmt werden.
Um dieses Problem zu lösen, wurde ein Projekt durchgeführt, welches die gängigen Techniken PCR (Polymerase chain reaction, auf deutsch: Polymerase-Kettenreaktion), Sequenzierung (Auslesen bestimmter DNA-Sequenzen) und Restriktionsverdau (Zerteilen der DNA mithilfe von Enzymen) nutzt, um auch Reste von Insekten bestimmen zu können.
Hierbei wird die mitochondriale DNA extrahiert und ein bestimmter Teil dieser Sequenz, der für jedes Insekt einmalig ist, ausgelesen.
Diese etwa 658 bp (Basenpaare) lange Sequenz kann mit Datenbanken abgeglichen werden und so zu einem Ergebnis führen.
Statt des Abgleichs mit Datenbanken kann auch ein Eins-zu-eins-Vergleich des unbekannten mit einem bekannten Insekt erfolgen, indem die DNA mittels Enzymen in definierte Stücke zerteilt und dann nebeneinandergestellt wird. Dies gewährt eine gewisse Unabhängigkeit, da auch ein Arbeiten ohne Datenbanken möglich ist.
Diese Techniken kamen im Fachbereich Schädlingsbekämpfung des LAVES zum Einsatz. Dieser Prozess ist aufwendig und wurde im Rahmen eines mehrjährigen Projekts durchgeführt.
Es konnten erfolgreich Insekten per Sequenzierung bestimmt werden, zum Beispiel Tribolium castaneum, Pheidole pallidula oder Lucilia silvarum.
Nach der Beendigung des Projektes ist die Technik am Lebensmittel- und Veterinärinstitut in Oldenburg etabliert worden.
 Bildrechte: ©LAVES/Beinke
Bildrechte: ©LAVES/BeinkeBeispiel Pheidole pallidula (Dickkopfameise)
Die Dickkopfameisen sind leicht an ihren dickköpfigen Arbeiterinnen zu erkennen. Bei der vom LAVES untersuchten Probe fehlten diese Arbeiterinnen. Es standen nur normale Arbeiterinnen und eine Königin für die Untersuchung zur Verfügung, daher bot sich die Verifizierung per Sequenzierung an. Das Ergebnis war eindeutig und eignete sich aufgrund der Schnittstelle gut zum Restriktionsverdau mit dem Enzym RsaI (sprich: R-S-A 1).
 Bildrechte: ©LAVES/Beinke
Bildrechte: ©LAVES/Beinke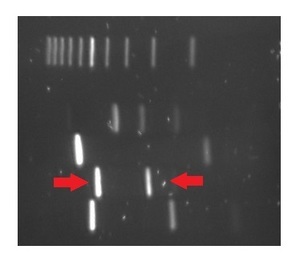 Bildrechte: ©LAVES/Beinke
Bildrechte: ©LAVES/BeinkeDie Ladder
Die Ladder (Leiter) dient als Maßstab und besteht aus verschieden langen DNA-Fragmenten, die aufgrund ihrer definierten Größe auch eine definierte Wanderungsgeschwindigkeit im Agarosegel haben. Die Ladder wandert mit den Proben mit und zeigt so an, wie groß die einzelnen Fragmente sind.



 English
English